最简 nnUNet 训练
准备数据
获得数据
已经将大家分割的图像进行了格式整理和一些预处理,并将其存放在如下目录下

volume和label分别是原图和大家手动分割好的标签图

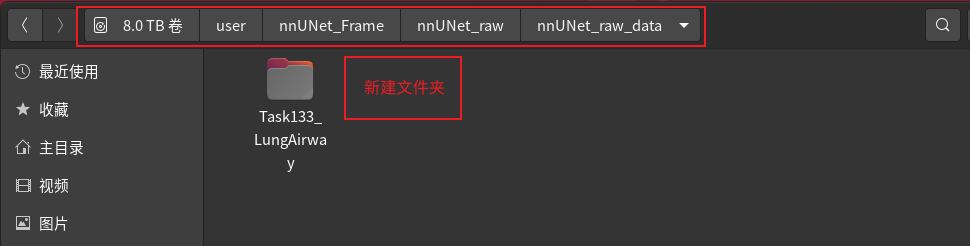
创建分割任务文件夹

然后在nnUNet_raw_data下新建文件夹 TaskXXX_任务名。 XXX是任务序号(大于100的数,前100被占用了,不如大家就取100 + 自己组的id,比如第21组就取名Task121_任务名),任务名取一个好记的英文名。
最后的文件结构如下
1
2
3
4
5
6
7
|
nnUNet_raw/nnUNet_raw_data/
├── Task101_BrainTumour
├── Task102_Heart
├── Task103_Liver
├── Task104_Hippocampus
├── Task105_Prostate
├── ...
|
然后在任务文件夹里构建如下结构
1
2
3
4
5
|
Task101_BrainTumour/
├── dataset.json
├── imagesTr
├── (imagesTs)
└── labelsTr
|
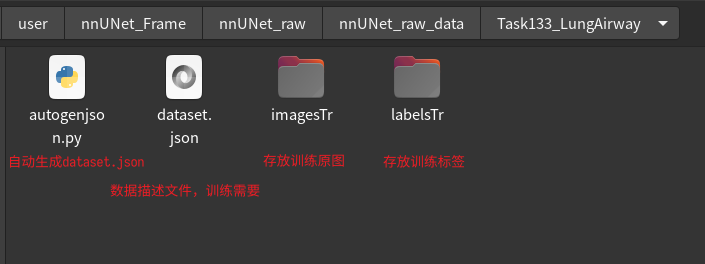
imagesTr 和 labelsTr 分别存放训练的原图和标签。imagesTs存放测试的原图(可以没有)。dataset.json 下面会说。旁边的Task133_LungAirway是一个范例,可以在根据这个进行学习和修改。
图像命名(可能是整个过程最难的环节)
imagesTr 和 labelsTr 中图像的命名也有要求。
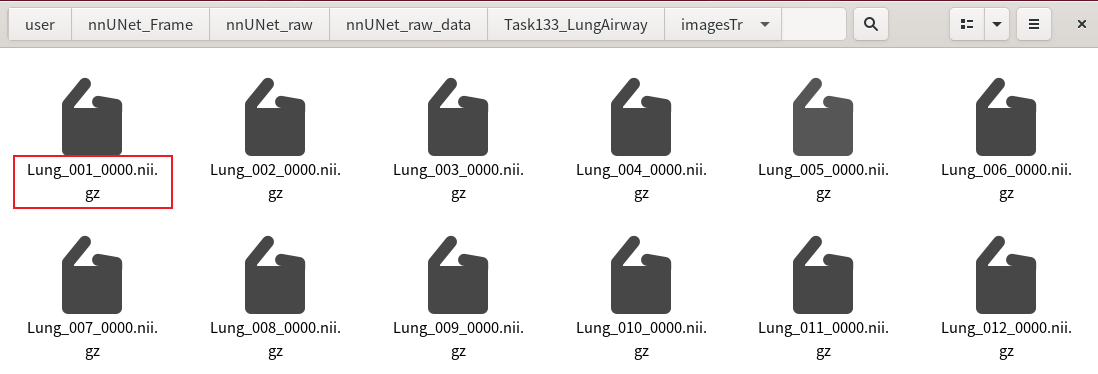
其中imagesTr(也就是原图)需要改成类似下图这样,而且必须是 .nii.gz 格式。建议写个 Python 脚本批量重命名并保存成 .nii.gz 格式(如果数量少,也可以手动修改)。

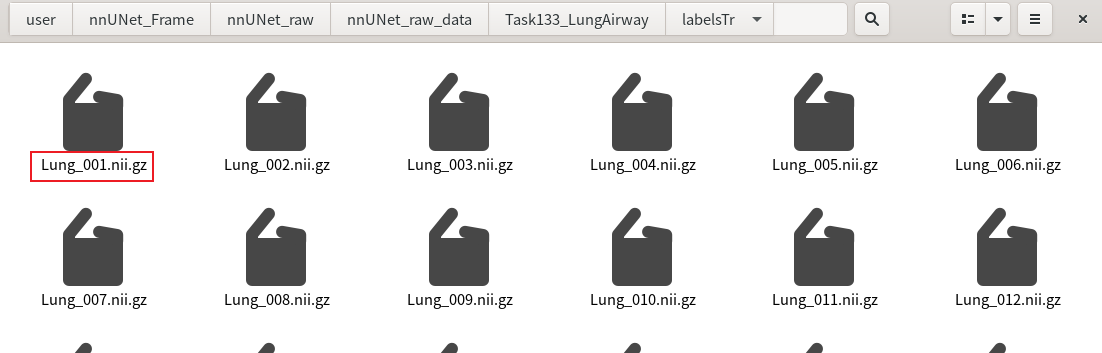
labelsTr(也就是标签图)需要改成如下样式,注意原图和标签图序号一一对应,只是原图后面带**_0000**,而标签图不带。

dataset.json 编写
上文说到这个任务文件夹应该包含一个 dataset.json 文件。下面是一个例子(可以在Task133里找到)
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
|
{
"description": "Task128_LungLobe",
"labels": {
"0": "0",
"1": "1"
},
"licence": "see challenge website",
"modality": {
"0": "CT"
},
"name": "Task128_LungLobe",
"numTest": 0,
"numTraining": 7,
"reference": "see challenge website",
"release": "0.0",
"tensorImageSize": "4D",
"test": [],
"training": [
{
"image": "./imagesTr/LungLobe_000.nii.gz",
"label": "./labelsTr/LungLobe_000.nii.gz"
},
{
"image": "./imagesTr/LungLobe_001.nii.gz",
"label": "./labelsTr/LungLobe_001.nii.gz"
},
{
"image": "./imagesTr/LungLobe_002.nii.gz",
"label": "./labelsTr/LungLobe_002.nii.gz"
},
{
"image": "./imagesTr/LungLobe_003.nii.gz",
"label": "./labelsTr/LungLobe_003.nii.gz"
},
{
"image": "./imagesTr/LungLobe_004.nii.gz",
"label": "./labelsTr/LungLobe_004.nii.gz"
},
{
"image": "./imagesTr/LungLobe_005.nii.gz",
"label": "./labelsTr/LungLobe_005.nii.gz"
},
{
"image": "./imagesTr/LungLobe_006.nii.gz",
"label": "./labelsTr/LungLobe_006.nii.gz"
}
]
}
|
按自己的任务的属性,修改上面的labels,numTest,numTraining等等信息。注意:这里image的文件名里没有 _0000 。但是修改这个json文件夹的工作量是比较大的,推荐写个 Python 脚本自动生成(当然如果只有几张图,手动改一改也是可以的)。下面是例子
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
|
from collections import OrderedDict
from batchgenerators.utilities.file_and_folder_operations import save_json
def main():
foldername = "Task128_LungLobe" #训练的名称
numTraining = 80 #训练集数量
numTest = 6 #测试集数量
numClass = 6 #分割的类别,如果不是多器官分割 则为2
json_dict = OrderedDict()
json_dict['name'] = foldername
json_dict['description'] = foldername
json_dict['tensorImageSize'] = "4D"
json_dict['reference'] = "see challenge website"
json_dict['licence'] = "see challenge website"
json_dict['release'] = "0.0"
json_dict['modality'] = {
"0": "CT", #模态
}
json_dict['labels'] = {i: str(i) for i in range(numClass)}
json_dict['numTraining'] = numTraining
json_dict['numTest'] = numTest
json_dict['training'] = [{'image': "./imagesTr/LungLobe_{:0>3d}.nii.gz".format(i),
"label": "./labelsTr/LungLobe_{:0>3d}.nii.gz".format(i)}
for i in range(numTraining)]
json_dict['test'] = ["./imagesTs/LungLobe_{:0>3d}.nii.gz".format(i)
for i in range(numTraining, numTraining+numTest)]
#上面的LungLobe可以换成自己的任务名
save_json(json_dict, "./dataset.json")
if __name__ == "__main__":
main()
|
需要发挥一点主观能动性,修改一下使之适应自己的任务。运行该脚本需要终端将目录切换到任务文件夹下,然后运行脚本。在终端输入以下命令

结果

训练
- 整理完数据,打开终端,并激活 nnUNet 环境,运行下面命令,
1
|
nnUNet_plan_and_preprocess -t XXX --verify_dataset_integrity
|
XXX 代替为你的任务序号。该命令进行预处理。(需要一定时间,耐心等待)
- 开始训练。
单卡训练:执行
1
|
nnUNet_train 3d_fullres nnUNetTrainerV2 XXX 0
|
意思是对序号为XXX的任务进行第0折交叉验证,训练模式是3d的全像素。一共有五折交叉验证,所以最后一个数为0-4。